Utente:Annamaria.dmr/frammento di Okazaki
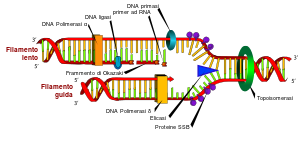
Un frammento di Okazaki è un breve frammento di DNA (composto da circa 1000-2000 nucleotidi) sintetizzato, nei procarioti, dalla DNA polimerasi III durante la replicazione del filamento lento del DNA, e da un innesco di RNA.
Poiché la sintesi del DNA può avvenire solo in direzione 5’-3’, solo uno dei due filamenti, detto "leading strand", può essere sintetizzato in maniera continua, utilizzando un unico innesco. L’altro filamento, invece, detto "lagging strand", deve essere sintetizzato in direzione opposta, sotto forma di piccoli frammenti
Il modello di questo meccanismo di replicazione del DNA discontinuo fu postulato da Reiji Okazaki, il quale dimostrò la presenza di questi frammenti, che da lui presero in seguito il nome.
La sintesi di ogni frammento di Okazaki inizia vicino alla forca replicativa a partire da un innesco (in inglese primer) da parte dell'enzima DNA polimerasi III. Il primer verrà poi rimosso dall'enzima RNAsi H e sostituito con DNA da parte dell'enzima DNA polimerasi I. Il frammento viene poi legato al frammento successivo da parte dell'enzima DNA ligasi tramite un legame fosfodiesterico per creare una catena di DNA continua.
Esperimenti di Okazaki modifica
Rejj Okazaki, nel 1968, insieme con i suoi collaboratori, condusse alcuni classici esperimenti che dimostrarono l’esistenza di questi corti filamenti di DNA:
- si allestirono delle colture del batterio Escherichia coli, che si trovavano in una fase di attiva sintesi di DNA;
- queste colture vennero sottoposte a brevi esposizioni (5 secondi) di timidina triziata, 3H-timidina, che era incorporata nel DNA nascente (pulse);
- si aggiungeva, quindi , timidina non radioattiva (cold chase);
- subito dopo il DNA veniva isolato e denaturato in soluzione alcalina;
- la soluzione veniva centrifugata in gradiente di saccarosio, che separava i filamenti di DNA in base al peso molecolare;
- le varie bande erano, quindi, analizzate per la presenza di radioattività.
L’esperimento fu ripetuto più volte, allungando i periodi delle fasi pulse e/o chase.
Si osservò che, quando l’esposizione al nucleotide marcato era molto breve (5-10 secondi), la maggior parte della radioattività era localizzata in corti frammenti di DNA, della lunghezza di 1000-2000 nucleotidi. Allungando il tempo d’esposizione (pulse), invece, aumentava la frazione di DNA marcato con più elevato peso molecolare. Lo stesso risultato si otteneva, quando si prolungava il periodo della cold chase, prima dell’isolamento del DNA.
Da queste osservazioni si dedusse che il DNA era sintetizzato sotto forma di piccoli frammenti nella prima fase, che poi erano saldati per formare filamenti più lunghi. Si dimostrò che era l’enzima ligasi a saldare i framenti di Okazaki, infatti, lo stesso esperimento condotto con mutanti ligasi-negativi (batteri che non possedevano una forma funzionale dell’enzima) non produceva filamenti radioattivi con elevato peso molecolare.
In seguito, la sintesi dei frammenti di Okazaki è stata evidenziata anche negli eucarioti e nei virus.
-->