Citoscheletro (Procarioti)
Con il termine citoscheletro procariotico ci si riferisce collettivamente a tutti i filamenti strutturali presenti nei procarioti.
Una volta si pensava che le cellule procariotiche non avessero citoscheletro, ma lo sviluppo delle tecnologie di microscopia e di biologia strutturale hanno portato alla scoperta di filamenti in queste cellule nei primi anni del 1990.[2]
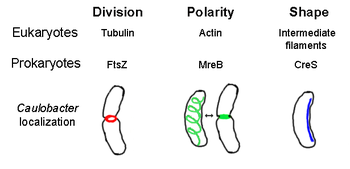
Oltre ad analoghi proteici presenti nella maggior parte dei citoscheletri degli eucarioti, nei procarioti sono state scoperte proteine citoscheletriche senza omologhi eucariotici ancora conosciuti.[3][4][5][6] Gli elementi strutturali del citoscheletro giocano ruoli fondamentali, nei vari procarioti nella: divisione cellulare; protezione; determinazione della forma; determinazione della polarità.[7][8]
FtsZ - Omologo funzionale della Tubulina
modificaFtsZ è stato il primo elemento del citoscheletro dei procarioti ad essere identificato.
Forma una struttura ad anello filamentosa localizzata al centro della cellula, denominata "Z-Ring" (Anello Z) che si contrae durante la divisione cellulare, simile all'anello actino-miosinico degli eucarioti. L'anello Z è una struttura altamente dinamica composta da numerosi fasci di protofilamenti che si estendono e contraggono, sebbene non sia chiaro il meccanismo che si cela alla base della contrazione dell'anello e del numero di protofilamenti coinvolti.
FtsZ agisce come una proteina "organizzatrice" ed è necessaria per la divisione cellulare. È il primo componente del setto durante la citocinesi e recluta altre proteine della divisione cellulare verso il sito di divisione.[9]
Sebbene questa somiglianza funzionale all'actina, FtsZ è considerato l'omologo della tubulina eucariotica. Nonostante confronti della struttura primaria di FtsZ e della tubulina rivelino una scarsa correlazione, le loro strutture tridimensionali sono decisamente simili. Pertanto, come la tubulina, FtsZ monomerica si lega al GTP e polimerizza con altri monomeri di FtsZ con l'idrolisi di GTP in un meccanismo simile alla dimerizzazione della tubulina.[10].
Poiché FtsZ è fondamentale per la divisione cellulare nei batteri, questa proteina è un bersaglio sfruttato durante la scoperta di nuovi antibiotici.[11] Esistono diversi modelli e meccanismi che regolano la formazione dell'anello Z.
Uno dei modelli della formazione dell'anello Z venne proposto da Rashid, Aijaz e Sing, Perminder[12]. In base a questo modello i regolatori positivi e negativi di FtsZ si assemblano a partire da un gradiente all'interno della cellula batterica. Il gradiente è tale che la concentrazione critica di FtsZ necessaria per l'assemblaggio di FtsZ sia iminima alla posizione mediale della cellula. Pertanto l'anello Z ed infine il complesso del divisoma cellulare si forma al centro della cellula.
MreB - Omologo funzionale dell'Actina
modificaMreB è una proteina batterica che si ritiene sia un analogo all'actina eucariotica. MreB e l'actina posseggono una scarsa somiglianza della loro struttura primaria, ma sono molto simili nella loro struttura tridimensionale e nel processo di polimerizzazione dei filamenti.
Quasi tutti i batteri non sferici dipendono da MreB per la determinazione della loro forma.
MreB si assembla in una rete elicoidale di strutture filamentose appena al disotto della membrana cellulare, coprendo l'intera lunghezza della cellula[13] MreB determina la forma della cellula modulando la posizione e l'attività di enzimi che sintetizzano peptidoglicani e agendo come un filamento rigido al di sotto della membrana cellulare che esercita una pressione verso l'esterno per scolpire e sostenere la cellula MreB inoltre si condensa dalla sua rete elicoidale e forma un anello rigido a livello del septum in Caulobacter crescentus appena prima della divisione cellulare, in un meccanismo che si pensa localizzare il suo septum off-center[14] MreB è inoltre importante per la determinazione della polarità dei batteri polari, ed è responsabile per il posizionamento corretto di almeno quattro proteine polari diverse in C. crescentus.
CreS (Crescentina) - Omologo funzionale dei filamenti intermedi
modificaCrescentina (codificata al gene creS) è un analogo dei filamenti intermedi (IFs) eucariotici. A differenza di tutte le altre relazioni di somiglianza descritte in questo articolo, la crescentina ha un'omologia di struttura primaria piuttosto considerevole rispetto alle proteine dei IFs, oltre che ad un'elevata somiglianza della struttura tridimensionale.
La sequenza di CreS ha infatti:
- un 25% di uguaglianza e un 40% di somiglianza con la citocheratina 19;
- un 24% di uguaglianza e un 40% di somiglianza con la lamina A (nucleare),
Inoltre, i filamenti di crescentina hanno circa un diametro di 10nm e pertanto sono nel range di diametro ideali dei IFs degli eucarioti (8-15nm).[15]
Crescentina forma un filamento continuo da un lolo all'altro lungo la parte interna e concava del batterio a forma di mezzaluna Caulobacter crescentus. Sia MreB che la crescentina sono necessarie per l'esistenza di C. crescentus nella sua forma caratteristica: si crede che MreB sposti la forma della cellula in un bastoncello e la crescentina modifichi la forma in una mezzaluna
Crenactina
modificaLa Crenactina è un omologo dell'actina esclusivo del regno degli archei Crenarchaeota scoperto nell'ordine dei Thermoproteales e Candidatus Korarchaeum.[16] Rispetto ad altri omologhi dell'actina, la crenactina presenta la più elevata somiglianza di sequenza con l'actina eucariotica.[17] La Crenactina è stata ben caratterizzata in Pyryobaculum calidifontis ed è stato dimostrato che ha alta specificità per l'ATP e il GTP. Le specie che contengono crenactina hanno tutte una forma rotonda o aghiforme ed in P. calidifontis la Crenactina ha mostrato formare strutture elicoidali che estendono la lunghezza della cellula, suggerendo un ruolo della crenactina nella determinazione della forma simile a quella di MreB in altri procarioti.[18]
Note
modifica- ^ vol. 120, DOI:10.1016/j.cell.2005.02.026, PMID 15766522, https://oadoi.org/10.1016/j.cell.2005.02.026. ]
- ^ vol. 354, DOI:10.1038/354161a0, PMID 1944597, https://oadoi.org/10.1038/354161a0.
- ^ vol. 128, DOI:10.1242/jcs.165563, PMID 25788699, https://oadoi.org/10.1242/jcs.165563.
- ^ vol. 287, DOI:10.1074/jbc.M112.341016, PMID 22514279, https://oadoi.org/10.1074/jbc.M112.341016.
- ^ vol. 397, DOI:10.1016/j.jmb.2010.02.010, PMID 20156449, https://oadoi.org/10.1016/j.jmb.2010.02.010.
- ^ vol. 194, DOI:10.1083/jcb.201102065, PMID 21859859, https://oadoi.org/10.1083/jcb.201102065.
- ^ vol. 70, DOI:10.1128/MMBR.00017-06, PMID 16959967, https://oadoi.org/10.1128/MMBR.00017-06.
- ^ vol. 75, DOI:10.1146/annurev.biochem.75.103004.142452, PMID 16756499, https://web.archive.org/web/20061117183040/http://www2.mrc-lmb.cam.ac.uk/SS/Lowe_J/group/PDF/annrev2006.pdf (archiviato dall'url originale il 17 novembre 2006).
- ^ vol. 7, DOI:10.1016/j.mib.2004.10.010, PMID 15556027, https://oadoi.org/10.1016/j.mib.2004.10.010.
- ^ vol. 20, DOI:10.1002/(SICI)1521-1878(199807)20:7<523::AID-BIES1>3.0.CO;2-L, PMID 9722999, <523::AID-BIES1>3.0.CO;2-L https://oadoi.org/10.1002/(SICI)1521-1878(199807)20:7<523::AID-BIES1>3.0.CO;2-L.
- ^ vol. 321, DOI:10.1126/science.1159961, PMID 18801997, https://oadoi.org/10.1126/science.1159961.
- ^ Critical Concentration Gradient Model of Bacterial Cell Division, su hdl.handle.net, Nature Precedings, 2010. URL consultato il 10 gennaio 2018.
- ^ vol. 301, DOI:10.1016/j.yexcr.2004.08.005, PMID 15501443, https://oadoi.org/10.1016/j.yexcr.2004.08.005.
- ^ vol. 101, DOI:10.1073/pnas.0402638101, PMID 15159537, https://oadoi.org/10.1073/pnas.0402638101.
- ^ vol. 115, DOI:10.1016/S0092-8674(03)00935-8, PMID 14675535, https://oadoi.org/10.1016/S0092-8674(03)00935-8.
- ^ vol. 80, DOI:10.1111/j.1365-2958.2011.07635.x, PMID 21414041, https://oadoi.org/10.1111/j.1365-2958.2011.07635.x.
- ^ vol. 4, DOI:10.1186/1745-6150-4-9, PMID 19245710, https://oadoi.org/10.1186/1745-6150-4-9.
- ^ vol. 112, DOI:10.1073/pnas.1511568112, PMID 26178194, https://oadoi.org/10.1073/pnas.1511568112.